聚合酶鏈反應(yīng) ( PCR )在現(xiàn)代分子生物學(xué)和分子醫(yī)學(xué)中產(chǎn)生了重大影響�����。因此,我們現(xiàn)在生活在 PCR 之后的時代���,因為現(xiàn)在許多診斷測試都使用 PCR����。下圖說明了用于 PCR 的核酸模板的樣本來源以及衍生分析方法的樣本��。
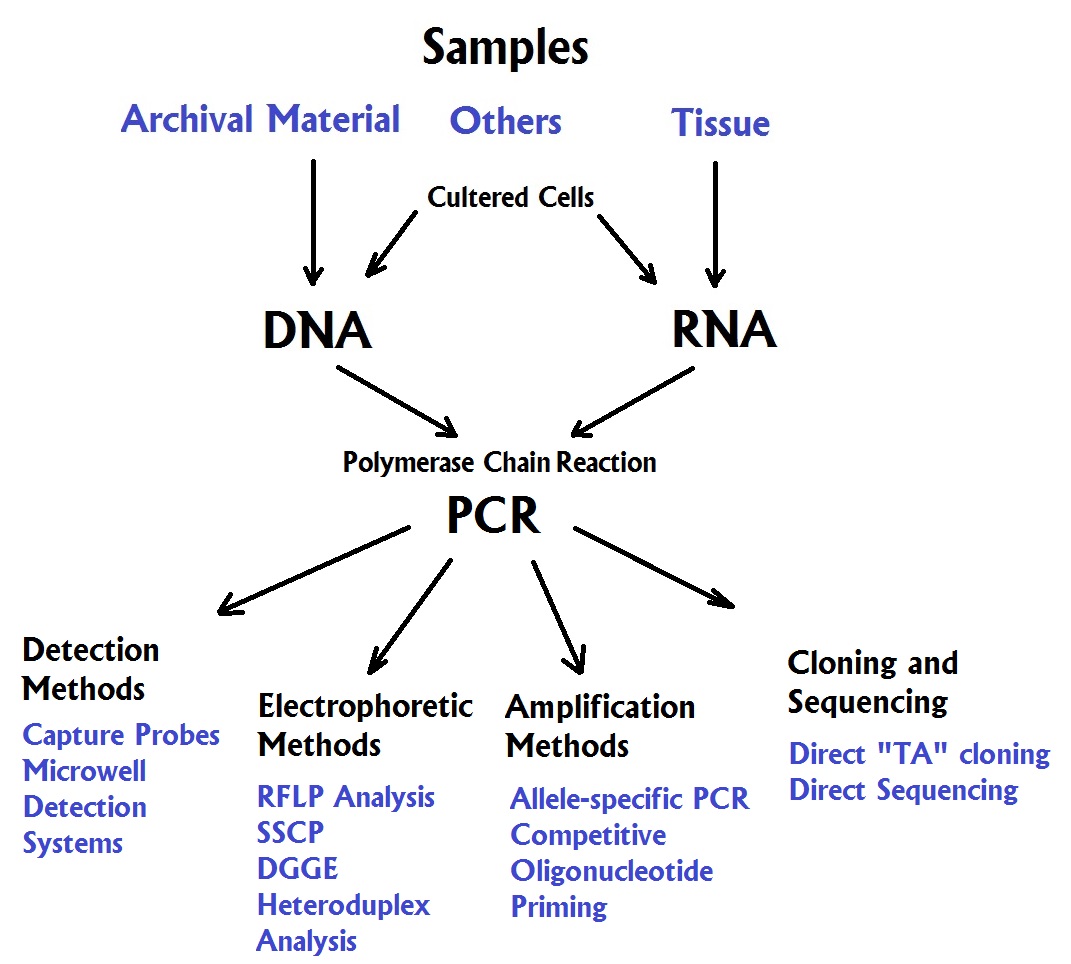
可使用 DNA 或 cDNA (RNA) 進行 PCR 分析����。有多種來源可用于制備核酸模板,包括法醫(yī)樣品��。PCR 現(xiàn)在形成了許多分析技術(shù)的基礎(chǔ)�。大多數(shù)方法利用目標序列的差異擴增,但其他方法可能涉及 PCR 產(chǎn)物分析的高級應(yīng)用��。
聚合酶鏈式反應(yīng) (PCR) 是一種允許在實驗室中使用現(xiàn)成的試劑對一段 DNA 進行多次拷貝的技術(shù)�����。在反應(yīng)過程中�,拷貝數(shù)呈指數(shù)增加�。 因此���,可以在幾個小時內(nèi)制作超過 1000 億份 DNA 片段���。 DNA聚合酶和合成寡核苷酸的可用性使這項技術(shù)成為可能。PCR 現(xiàn)在已成為克隆的替代方法���,因為它允許擴增復(fù)雜混合物中的特定序列�。此外�����,PCR 結(jié)合測序技術(shù)可以特異性���、快速和高靈敏度地鑒定 DNA 樣品中的突變等位基因���。
PCR 技術(shù)允許從少量起始材料中特異性擴增目標 DNA 序列。在早期版本的 PCR 中�,使用了的 Klenow 片段 大腸桿菌 DNA 聚合酶 I 。然而��,Klenow 片段不是 熱穩(wěn)定 . 引入熱穩(wěn)定的 DNA 聚合酶,例如在 Thermus 中發(fā)現(xiàn)的 DNA 聚合酶 水生動物 ��,導(dǎo)致 PCR 方法發(fā)展的重大技術(shù)突破����。
PCR 可以根據(jù)技術(shù)的使用方式分為不同的組或方法�����。 但是�����,所有這些方法都遵循相同的基本步驟和原則����。

圖 1:TAQ 聚合酶模型。
研究人員用與聚合酶結(jié)合的平端雙鏈 DNA 解析了 Taq 聚合酶的共晶結(jié)構(gòu) 活性位點 裂縫����。 該結(jié)構(gòu)表明 DNA 既不彎曲也不穿過大的聚合酶裂縫。結(jié)合 DNA 的結(jié)構(gòu)構(gòu)象介于 B 和 A 形式之間�。 結(jié)構(gòu)模型顯示寬的小溝允許進入蛋白質(zhì)側(cè)鏈。 側(cè)鏈在平端與嘌呤的 N3 和嘧啶的 O2 氫鍵連接���。
DNA聚合酶催化長多核苷酸鏈的合成�����。 在原始親本 DNA 鏈的存在下�,合成從單體脫氧核苷酸三磷酸開始。DNA 鏈用作合成新的互補 DNA 鏈的模板����。合成沿 5' 到 3' 方向進行。 聚合從脫氧核苷三磷酸的 5' α-磷酸酯到正在生長的 DNA 鏈的 3' 末端羥基發(fā)生�����。 DNA 聚合酶需要一小段 DNA 與互補序列退火���。 該 DNA 序列或 寡核苷酸 稱為引物�����,因為需要它來引發(fā)合成���。
聚合酶鏈式反應(yīng)或 PCR 是由 Kary B. Mullis 于 1983 年發(fā)現(xiàn)、構(gòu)思或發(fā)明的�。據(jù)他說��,他在月光下的加利福尼亞北部開車時偶然發(fā)現(xiàn)了這種反應(yīng)�。 此外��,正如他所指出的�����,該反應(yīng)允許在幾個小時內(nèi)從單個 DNA 分子生成多達 1000 億個類似的 DNA 分子�。 該反應(yīng)可以在試管中進行���,但也需要一些試劑和熱源�。 PCR 反應(yīng)的引入使分子生物學(xué)家的生活變得更加輕松����,使他們能夠生產(chǎn)盡可能多的 DNA。 該技術(shù)以驚人的速度傳播到整個生物科學(xué)領(lǐng)域����。 然而,由于反應(yīng)涉及熱循環(huán)�����,最終改進的 PCR 技術(shù)中最重要的部分是使用熱穩(wěn)定的 DNA 聚合酶。 最初從提取的聚合酶棲熱菌中水生動物 現(xiàn)在幾乎用于所有 PCR 反應(yīng)�����。 聚合酶鏈反應(yīng)已成為分子生物學(xué)領(lǐng)域的終極改變游戲規(guī)則的技術(shù)�����。
聚合酶鏈反應(yīng)的特點
* 聚合酶鏈式反應(yīng) (PCR) 選擇性地擴增目標 DNA 分子�����。
* 它允許在熱變性����、引物退火和引物延伸的重復(fù)循環(huán)中延伸短的單鏈合成寡核苷酸、引物����。
* PCR 是一個循環(huán)過程,其中一系列步驟一遍又一遍地重復(fù)���。
* 雙鏈 DNA 片段通過稱為變性的溫和加熱分離成單鏈����。
* 將短的合成寡核苷酸引物與四種脫氧核糖核苷酸三磷酸 dATP、dGTP�、dCTP、dTTP 和 DNA 聚合酶(現(xiàn)在通常是 Taq DNA 聚合酶)一起添加到反應(yīng)混合物中�。
輝駿生物PCR實驗服務(wù)流程
1.引物設(shè)計與合成;
2.基因模板制備:RNA提取和反轉(zhuǎn)錄�����,或DNA提?��?;
3.qPCR預(yù)實驗:檢測目的基因和內(nèi)參基因的擴增情況��;
4.qPCR正式實驗:全部樣本做qPCR�����,每個樣本設(shè)置三復(fù)孔�;確定樣本擴增產(chǎn)物單一��,內(nèi)參基因CT值正常�����;
5. 數(shù)據(jù)分析和出具報告:按照客戶要求計算樣本間的目的基因表達差異,并作柱形圖���。
參考
1. Eom SH, Wang J, Steitz TA.; Structure of Taq polymerase with DNA at the polymerase active site. Nature. 1996 Jul 18; 382(6588):278-81.
2. Faloona, F., Weiss, S., Ferre, F., and Mullis, K. 1990. Direct detection of HIV sequences in blood high-gain polymerase chain reaction [abstract]. In: 6th International Conference on AIDS, University of California, San Francisco: San Francisco (CA). Abstract 1019.
3. Gelfand, D.H. and White, T.J. 1990. Thermostable DNA polymerases. In: PCR Protocols: A Guide to Methods and Applications. Innis, M.A., Gelfand, D.H., Sninsky, J.J., and White, T.J., eds. San Diego: Academic Press. 129–141.
4. Holland, P.M., Abramson, R.D., Watson, R., and Gelfand, D.H. 1991. Detection of specific polymerase chain reaction product by utilizing the 5′→3′ exonuclease activity of Thermus aquaticus DNA polymerase. Proc. Natl. Acad. Sci. USA 88:7276–7280.
5. Innis, M.A., Myambo, K.B., Gelfand, D.H., and Brow, M.A. 1988. DNA sequencing with Thermusaquaticus DNA polymerase and direct sequencing of polymerase chain reaction-amplified DNA. Proc. Natl. Acad. Sci. USA 85:9436–9440.
6. Mullis K. In Methods In Enzymology, Vol.155, 335, 1987.
7. Kary B. Mullis; The Unusual Origin of the Polymerase Chain Reaction. SCIENTIFIC AMERICAN April 1990. 56-65.














 實驗熱線:4006991663
實驗熱線:4006991663





